Name des Teilnehmers: Maximilian Harl
Beschreibung des IT-Forschungsprojekts: Wirkstoffdesign, insb. Medikamentenentwicklung, ist im Allgemeinen sehr resourcen- und zeitintensiv mit Kosten bis zu 2.6 Milliarden Euro und Zeithorizonten bis zu 20 Jahren. Aus diesem Grund wurde in mehreren Studien der Nutzen von verschiedenen Verfahren des Machine Learnings für das Wirkstoffdesign untersucht und es konnte aufgezeigt werden, dass diese, im Vergleich zu klassischen Verfahren, sowohl schnell als auch relativ genau viele Eigenschaften von Molekülen vorhersagen können [1, 2].
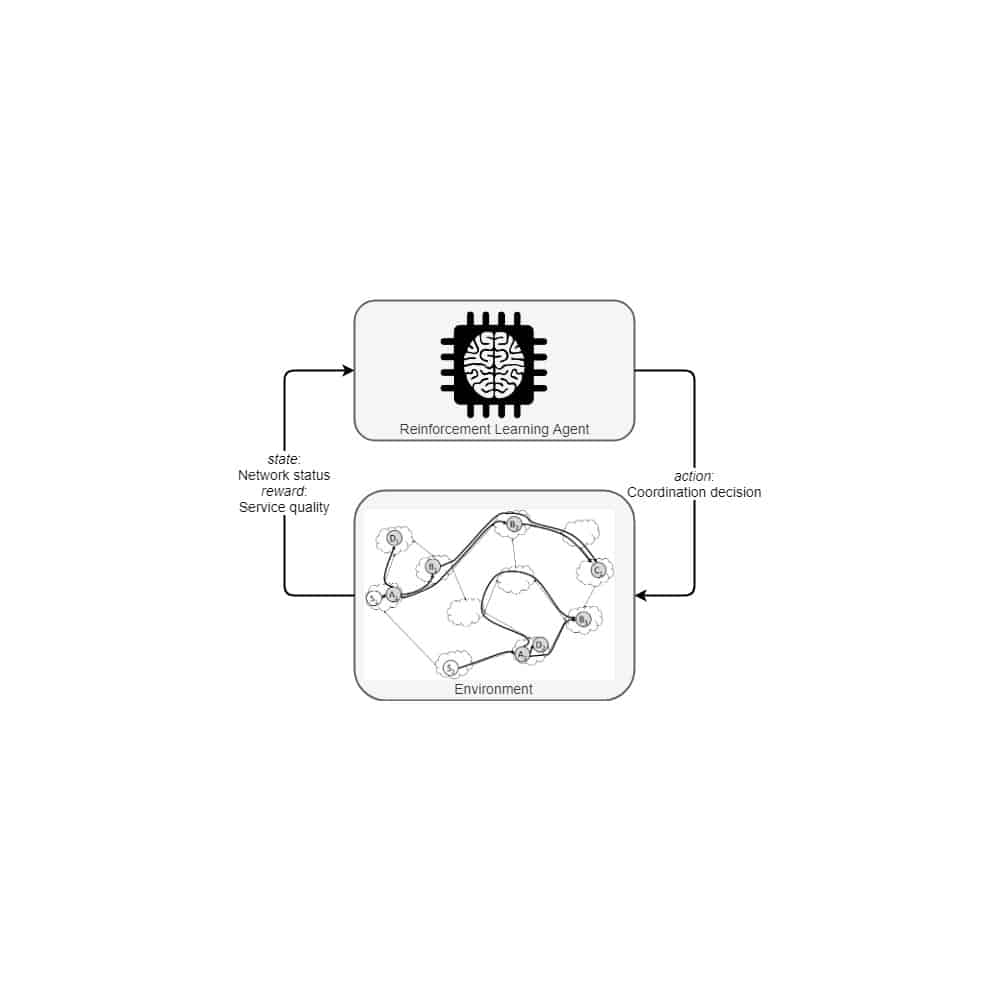
Dieses Forschungsprojekt beschäftigt sich mit der Untersuchung von Reinforcement Learning Verfahren im Wirkstoffdesign und Prozessmanagement (insb. Process Mining). Bei diesen Verfahren werden mögliche Entscheidungen mit Abschätzungen ihrer Auswirkungen analysiert um approximativ ein Verhalten zu bestimmen, welches das gewünschte Ziel verfolgt. Im Falle von Wirkstoffdesign sind dies gewünschte physiochemische oder biologische Eigenschaften. Mit den kontemporären Fortschritten im Deep Learning kommt es auch zu einer Renaissance des Reinforcement Learning, vor allem in Kombination beider Techniken. In dem Kontext dieses Projektes bietet Reinforcement Learning mehrere Vorteile, vorangestellt, dass die Modifizierungen von Molekülgraphen als Grundlage für Reinforcement Learning Verfahren verwendet werden können, wodurch ebenfalls nur chemisch valide Veränderungen vorgenommen werden [3].
Dieses Projekt wird untersuchen, wie Moleküle für Reinforcement Learning Verfahren optimal repräsentiert und wie Belohnungsfunktionen für das Wirkstoffdesign zielführend formuliert werden können. Des weiteren können diese (Molekül)graphen im Kontext des Prozessmanagement (insb. Process Mining) zur Modellierung von Geschäftsprozessen verwendet werden. Aus diesem Grund wird ebenfalls untersucht, welches Potenzial Methoden des Wirkstoffdesigns in der Domäne des Prozessmanagements (insb. Process Mining) haben und wie diese in beiden Domänen angewendet werden können.
Software Campus-Partner: FAU & Merck
Umsetzungszeitraum: 01.03.2021 – 28.02.2022
[1] Elton, D. C., Boukouvalas, Z., Fuge, M. D., & Chung, P. W. (2019). Deep learning for molecular design—a review of the state of the art. Molecular Systems Design & Engineering, 4(4), 828-849.
[2] Zhavoronkov, A., Ivanenkov, Y. A., Aliper, A., Veselov, M. S., Aladinskiy, V. A., Aladinskaya, A. V., … & Aspuru-Guzik, A. (2019). Deep learning enables rapid identification of potent DDR1 kinase inhibitors. Nature biotechnology, 37(9), 1038-1040.
[3] Zhou, Z., Kearnes, S., Li, L., Zare, R. N., & Riley, P. (2019). Optimization of molecules via deep reinforcement learning. Scientific reports, 9(1), 1-10.